近日,我校生物反应器工程全国重点实验室王启要教授课题组利用人工智能技术预测鉴定病原菌毒力效应蛋白方面取得新进展。该工作以“Contrastive-learning of language embedding and biological featuresfor cross modality encoding and effector prediction”为题在线发表在Nature Communications。
效应蛋白是一类由细菌病原微生物分泌到环境以及宿主细胞内的关键毒力蛋白,可以通过干扰宿主免疫信号通路、调控细胞代谢、协助摄取营养等方式促进病原菌感染。鉴定效应蛋白以及进行其分子机制研究对解析病原菌-宿主免疫互作机制和抗感染药物靶点开发具有重要意义。然而,许多效应蛋白在功能和蛋白质序列特征上具有多样性,使得基于序列保守性的预测方法较难有效预测未知效应物,而基于实验的筛选方法则存在着通量低和时间成本高等问题。为此,研究团队开发了人工智能赋能的蛋白质功能预测新方法,利用蛋白质语言模型进行效应蛋白预测鉴定,以突破传统技术路线的局限性。
近年来,该课题组团队以水产病原杀鱼爱德华氏菌的III/VI型分泌系统效应蛋白为研究对象,建立了基于转录组学、蛋白质组学分析的分泌系统效应蛋白筛选技术平台,成功鉴定到多个新型效应蛋白(Virulence 2017, 8: 1355-1377; CommunicationsBiology 2024,7: 162)。在上述基础上,研究团队结合预训练蛋白质语言模型和对比学习技术构造了名为CLEF的神经网络模型。该模型能够将语言模型对蛋白质序列编码与其他的生物学特征对齐,在现有模型基础上有效提升神经网络对效应蛋白的预测性能。利用该模型,课题组成功对杀鱼爱德华氏菌中潜在的未知效应蛋白进行预测,并通过实验成功鉴定12个新型III型分泌系统效应蛋白和11个VI型分泌系统效应蛋白。
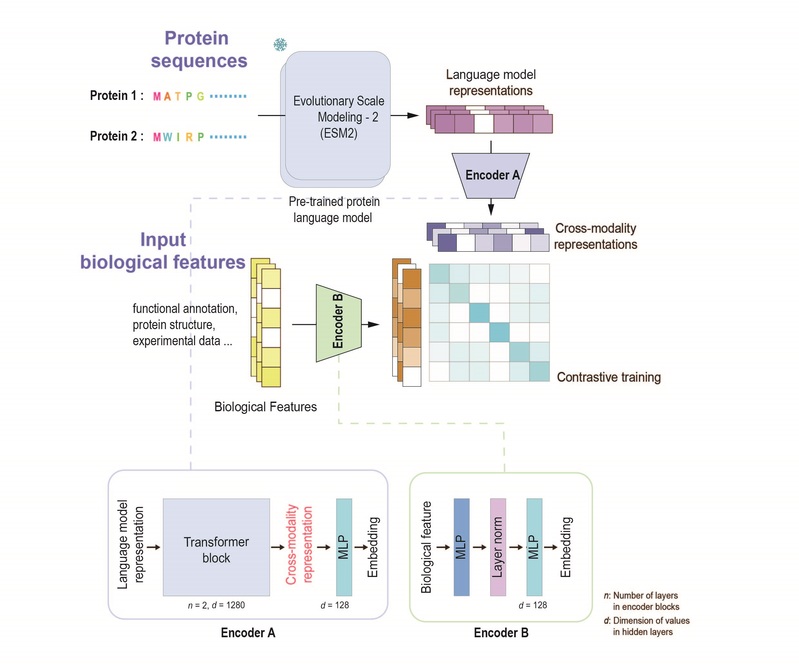
图片说明:CLEF的对比学习流程和神经网络架构
上述成果实现了人工智能与生物学湿实验数据的深度融合,为病原菌毒力因子的大规模挖掘提供了高效技术平台。该研究不仅推动了病原菌效应蛋白的功能鉴定,更为复杂的病原菌效应蛋白-宿主免疫互作机制网络的解析奠定了技术基础。
我校生物工程学院彭越博士为本研究的第一作者,邵帅副教授为本研究的通讯作者,研究工作得到了生物工程学院王启要教授、张元兴教授和刘琴教授的大力支持。该研究工作得到了国家自然科学基金重点和面上项目、科技部重点研发计划和国家现代农业产业技术体系等项目的资助。
论文链接:https://doi.org/10.1038/s41467-025-56526-1






